初めに
(ヒト)ヘモグロビンは、赤血球に含まれる分子です。赤血球は肺の毛細血管で酸素を「捕まえ」て、血流に乗って身体の組織に移動し、組織では酸素を「離し」ます。つまり、酸素を肺から身体の組織へ運ぶ機能を有します。その機能をヘモグロビンの構造の側面から観察してみました。
ヘモグロビンには2つの構造が異なる状態があります。緊張した状態 (tens form, T型)と緩んだ状態(relaxed form, R型)です。周辺(環境)のpHが低い、CO2濃度が高い、2,3 BPG濃度が高い等の状況下では、ヘモグロビン分子がT型を取りやすく、その逆ではR型を取りやすいとされています。T型は酸素との親和性が低く、言い換えるならば酸素を放出しやすく、逆にR型では酸素との親和性が高く、酸素と結合しやすい状態です。T型を取りやすい環境は身体では組織の環境であり、R型を取りやすい環境は肺の毛細血管の環境ということになります。赤血球が肺で酸素を取り込み組織まで運ぶのは、ヘモグロビンが組織では酸素を放出し、肺の毛細血管では酸素と結合しやすいというヘモグロビンの性質によって実現されています。
生理的に存在する2つのヘモグロビンの状態のうち、ヘモグロビン分子が酸素と結合した状態のものをoxyhemoglobin、酸素と結合していないものをdeoxyhemoglobinと呼びます。ヘム鉄はそのいずれの状態でも2価のままです。oxyhemoglobinを「酸素化ヘモグロビン」とdeoxyhemoglobinを「脱酸素化ヘモグロビン」と呼びます。「酸化ヘモグロビン」「還元ヘモグロビン」と書かれている場合がありますが、私はこの呼び方には違和感を持っています。oxyhemoglobinでも、ヘム鉄が酸化されていないからです。これらの酸素化・脱酸素化の状態の他に、実はヘム鉄が酸化した3価の状態の物も存在していまして、「メトヘモグロビン」と呼ばれています。病的にメトヘモグロビンが増加した状態をメトヘモグロビン血症と呼びます。私としてはメトヘモグロビンという病的な状態で増加する分子を「酸化ヘモグロビン」と呼びたいところです。(ただし、そのように呼んでいるものはあまり目にしません。)
オゾンで処理した自己血を輸注する健康法(?)の様なものが報道されていましたが、オゾンのような反応性の高い酸素原子を含む分子が、ヘモグロビンと反応するとメトヘモグロビンを産生しないか心配です。実はそのような実験を行った結果が報告されていました。等容量(液体とガスとが)のヘモグロビンと4%以下のオゾンを短時間 (within few minutes) 反応させたところメトヘモグロビンがごくわずか増加したということでした1。環境にあるオゾンが人に作用した際のヘモグロビンの酸化を評価するような実験なのか、この文献で試験されていましたのは、オゾン濃度が最大4%、生体分子とオゾンが反応すると短期間にオゾン濃度が低下することを想定して、ヘモグロビンが直接オゾンと反応するであろう反応時間も短時間に設定して実験しています。(Within few minutes after rapid mixing of the equal gas‐liquid volumes, the ozone was consumed because by instantaneously reacting with biomolecules, generating reactive oxygen species (particularly hydrogen peroxide) having very short lifetime and lipid oxidation products. ) そうした実験系でもメトヘモグロビンはごくわずかに増加を示したようです。
という訳で?酸素化ヘモグロビンと脱酸素化ヘモグロビンの構造の差を見てみようと思いました。
方法
構造の情報はProtein Data Bankから
deoxyhemoglobin 2hhb.pdb (1)
oxyhemoglobin 1hhb.pdb2 (2)
α鎖1分子分のみ表示したい
(1) chainA + heme Aのみsave -> deoxyHb.pdb (3)
(2) chainA + heme A + o のみsave -> oxyHb.pdb (4)
(3) load (4) load -> (5)
作業中見やすくするように
(5) 両方のレイヤーについてside chainをかくす
(side chainのカラムのどこかを右クリック、これを両方のレイヤーで行う)
(6) OxyHb ピンク DeoxyHb 水色 O 赤 Oxyのヘム黄色 Deoxyのヘムマゼンタ
2種類の構造の位置を一致させて表示するために
(7) Fit メニュー:Magic Fit -> accept all parameters
(8) Fitメニュー: Generate Structure alignment
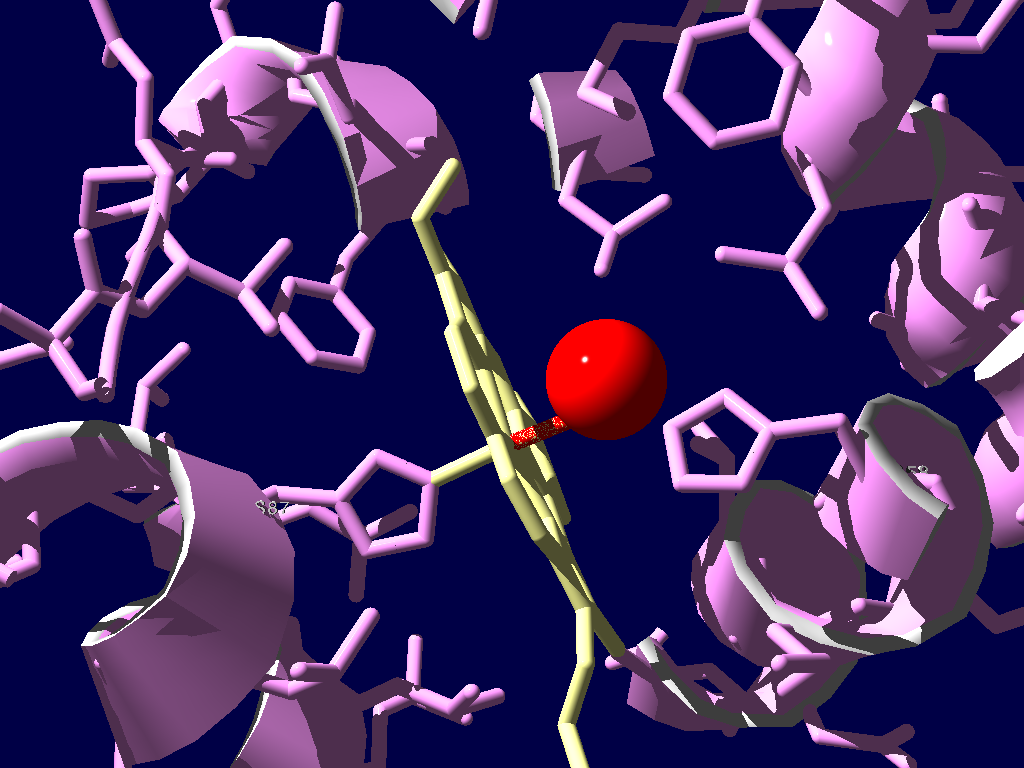
ヘムと相互作用するカギとなるヒスチジン残基
(9) サイドチェインを表示、His87, His58のラベルを表示
図を美しくしたい~レイトレーシング
(10) 見やすそうな角度で、PovRayへの出力ファイルを保存
結果
酸素化ヘモグロビン
脱酸素化ヘモグロビン

上記の2つを同じ位置に配置してみました

想うところ
赤血球が酸素を運ぶ際、鉄が錆(さび)ようとする(酸化されやすい)性質を利用して酸素を引き付けます。しかし、本当に錆びてしまったら酸素を必要な場所で放すことができませんので、引き付けるエサとして鉄を利用しつつ、その鉄に近づいた酸素をうまく制御している、というのがヘモグロビンの機能の本質的な部分であって、あまり強力な酸化作用はちょうど良い制御を逸脱するかもしれません。
前々職の開発にかかわった医薬品の副作用として、「メトヘモグロビン血症」がありました。メトヘモグロビン血症を診断した場合にはメチレンブルーを使用することになるのですが、その当時日本国内ではメチレンブルーが医療用医薬品として承認されておらず、ひどいメトヘモグロビン血症になったらどうするといいかという問いには非常に答えにくい状況でした。金魚等の白点病の治療薬としてはペット屋さんに行けばありますが、人に静脈注射できるような品質が担保されていません。医療上の必要性が高い未承認の医薬品という事で厚生労働省から働きかけがあり、第一三共さんが承認を取得して世に送り出してくださったという経緯があります。
造血幹細胞移植を行う際に無菌室を滅菌するのにオゾンを用いていたこともあり、オゾンは殺菌作用のある物質と認識しており、生体分子との反応性も高そうです。人にとっても高濃度曝露は避けるべきものと理解していました。そういう認識があったもので個人的には毒性が心配なのですが、それが健康法(「クレンジング」)として話題になっていて気になりましたので、どのように安全性が調べられたのか、この健康法を行うというクリニックのホームページをいくつか見ましたが方法がきちんと書かれていないようです。
References:
1.
Bocci V, Aldinucci C. Biochemical modifications induced in human blood by oxygenation-ozonation.
J Biochem Mol Toxicol. 2006;20(3):133-138.
[PubMed]
2.
Fermi G, Perutz M, Shaanan B, Fourme R. The crystal structure of human deoxyhaemoglobin at 1.74 A resolution.
J Mol Biol. 1984;175(2):159-174.
[PubMed]
 今回は他ユニットとのジョイントライブで、’たまごぶらす’は最後でした。仕事が終わってホールに入るとフルート2本とピアノのユニットが最後の曲を演奏していました。
今回は他ユニットとのジョイントライブで、’たまごぶらす’は最後でした。仕事が終わってホールに入るとフルート2本とピアノのユニットが最後の曲を演奏していました。 2組目の木管五重奏が始まる前の休憩の間に座席に案内されました。少し遅めに行ったという事でステージで言う上手側端の方の座席でした。流石に目の前のクラリネットの音ばかりが響く感じでしたが、このクラリネットの音が心地よくて’たまごぶらす’が始まる前から大満足です。吉田かなえさん、素晴らしい演奏ありがとうございます!
2組目の木管五重奏が始まる前の休憩の間に座席に案内されました。少し遅めに行ったという事でステージで言う上手側端の方の座席でした。流石に目の前のクラリネットの音ばかりが響く感じでしたが、このクラリネットの音が心地よくて’たまごぶらす’が始まる前から大満足です。吉田かなえさん、素晴らしい演奏ありがとうございます! たまごぶらすの曲は以下の通りでした。お得意のレパートリーでしたが、それぞれ以前より一段とこなれてきて楽しめました。
たまごぶらすの曲は以下の通りでした。お得意のレパートリーでしたが、それぞれ以前より一段とこなれてきて楽しめました。