R summary tools
はじめに
R summary toolsを使ってみました。データフレームのデータを簡単にサマライズしてくれます。
Rスクリプトです
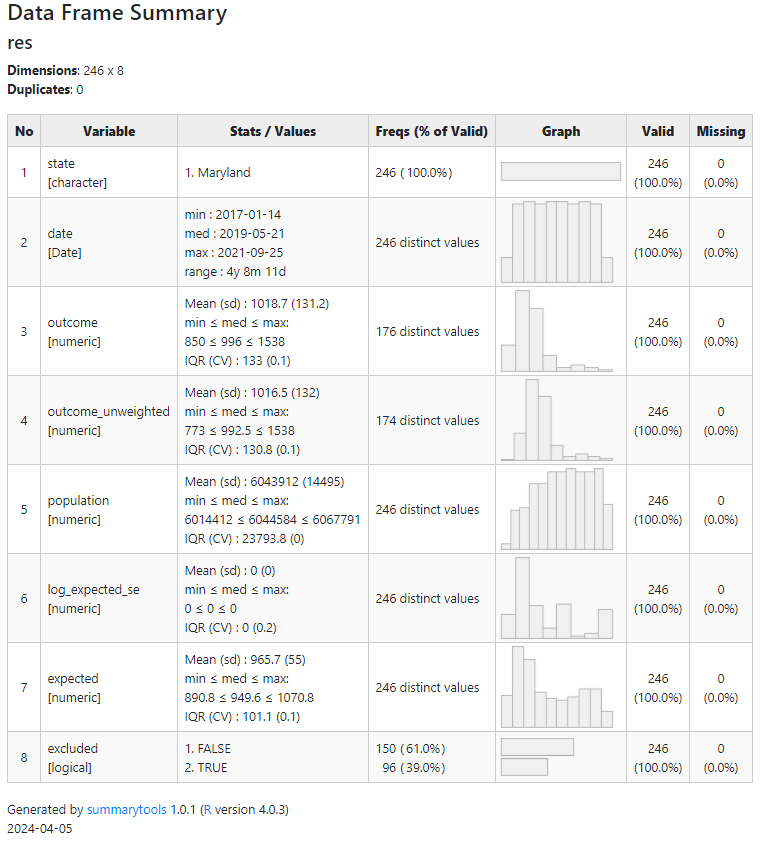
library(survival) #このパッケージに付属する lung というデータのサマリーを作成します library(summarytools) dfs <- summarytools::dfSummary(lung, max.distinct.values = 10) summarytools::view(dfs) #### 結果のdfsと言うデータフレームを保存する必要なければパイプで表示に送って summarytools::dfSummary(lung, max.distinct.values = 10) %>% summarytools::view()
解説です
このRスクリプトは、’survival’と’summarytools’という2つのライブラリを使って、’lung’というデータセットの概要を表示し、その概要を’dfs’というデータフレームに保存しています。 まず、’library(survival)’と’library(summarytools)’は、それぞれ’survival’と’summarytools’というライブラリを読み込むコマンドです。これらのライブラリは事前にインストールされている必要があります。 次に、’summarytools::dfSummary(lung, max.distinct.values = 10)’は、’lung’というデータセットの要約統計量を計算し、その結果をデータフレーム形式で返します。’max.distinct.values = 10’というオプションは、各列の異なる値が10以下の場合にはすべて表示し、それ以上の場合には最も頻繁に現れる10個の値のみ表示するという意味です。 このデータフレームは’dfs’という名前で保存され、’summarytools::view(dfs)’によって表示されます。 また、’summarytools::dfSummary(lung, max.distinct.values = 10) %>% summarytools::view()’という部分は、上記の2つの操作を1行で行うための別の書き方です。’%>%’はパイプ演算子と呼ばれ、左側の結果を右側の関数に渡します。この場合、’summarytools::dfSummary(lung, max.distinct.values = 10)’の結果が直接’summarytools::view()’に渡されて表示されます。
結果です