ゲノムワイド関連解析(GWAS)のマンハッタンプロット
アイキャッチ画像のような図を論文で見ていて、どうやって描いたもので何を意味するのか自分でもとデータから図を描きつつ見方を学ぼうという事で書籍を開きながら試してみました。その時のチョコっとした学びを書き留めた備忘録です。
#初心者によるやってみた系の記事
参考にした書籍
参考にしたのは「ゼロから実践する 遺伝統計学セミナー〜疾患とゲノムを結びつける」という本です。参考というか、このホームページに書いていることは、この本に書かれていることをたどっただけです。書籍にはCGYWINのインストール、plinkの使い方から、マンハッタンプロットまで丁寧に書かれていますので、この書籍を読めば私のホームページは見る価値があまりないかもしれません。
Rでのプロット
ここで、今回使用したRのスクリプトを掲載しようと思っていたのですが、そのスクリプトというのが上記書籍を購入したらダウンロードできるというデータの中にあるスクリプトで、ごくわずか変更(ファイル格納ディレクトリやデータファイル名を変更)した程度なので、さすがに掲載したらまずそうだと思いとどまり、別の例で示します。
CRANから、Rのパッケージqqmanをインストールしてあるとして次になります。
library(qqman) manhattan(gwasResults, chr="CHR", bp="BP", snp="SNP", p="P" )
gwasResultsはパッケージqqman付属のデータで、次のようにSNPの名称・染色体・位置・表現型と当該SNPとの関連性がないと仮定した場合の確率P値の4項目のデータが16470行続きます。
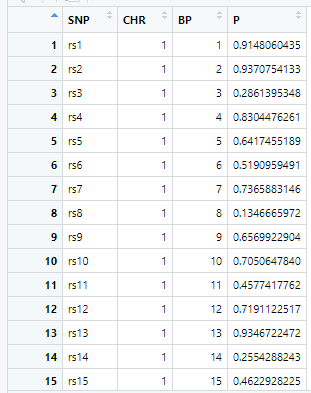
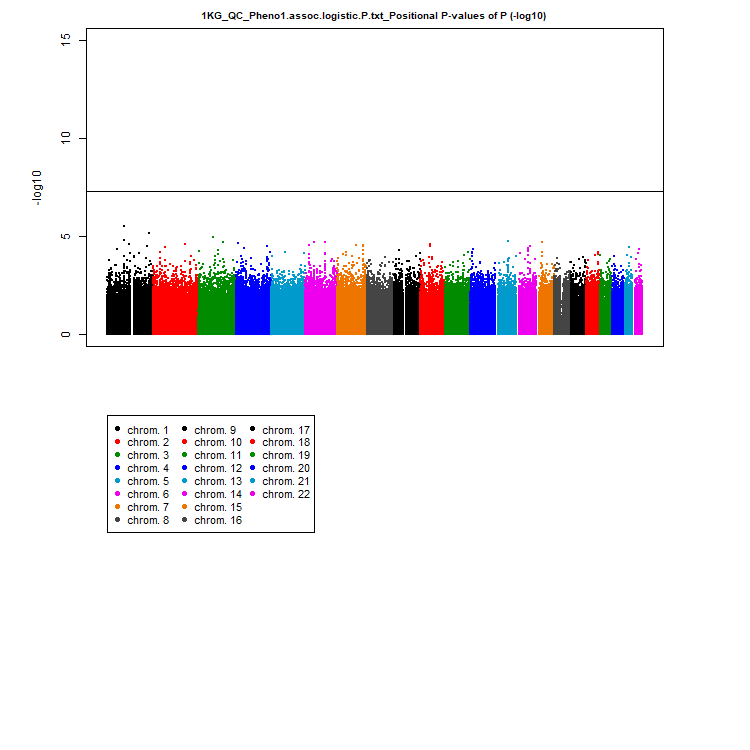
で、このスクリプトの結果は次の図のようになります。
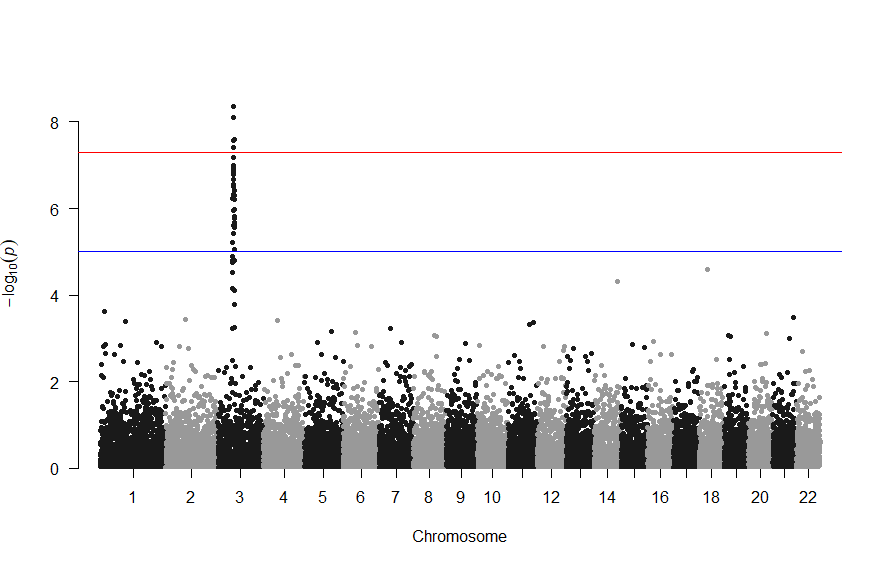
色が寂しいので、同じデータでウクライナを思わせるカラーで表現すると
# Load the library
library(qqman)
# Make the Manhattan plot on the gwasResults dataset
manhattan(gwasResults, chr="CHR", bp="BP", snp="SNP", p="P", col = c("#0057B7", "#FFDD00"))
次のようになります
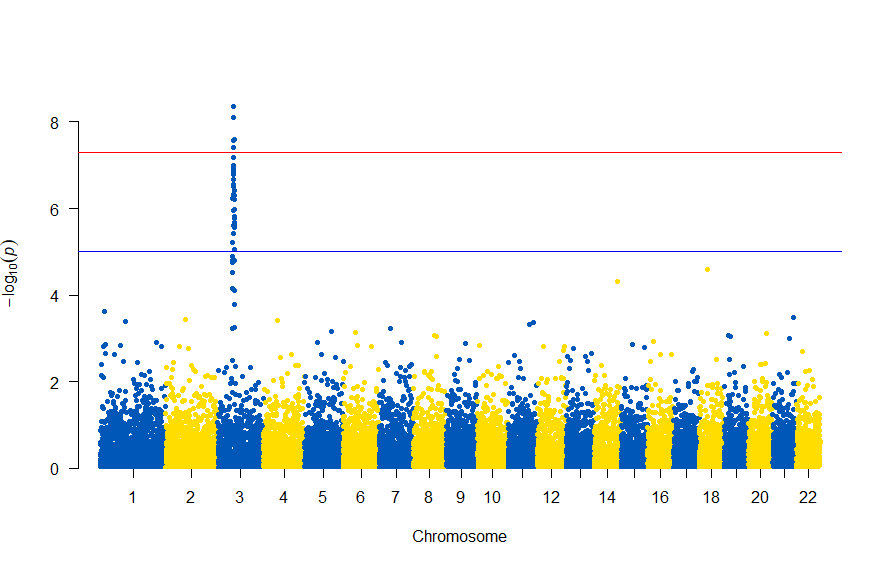
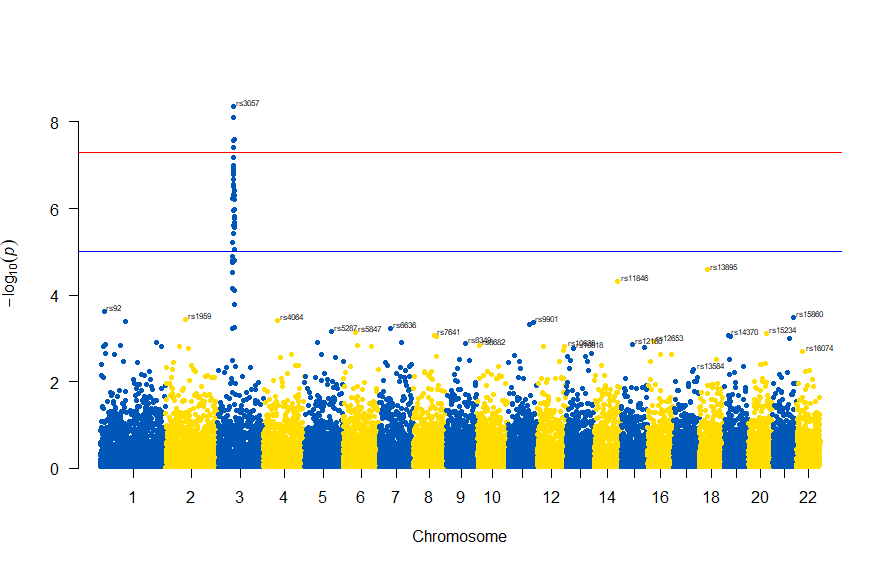
図の中のSNPを表示するには , annotatePval = 0.01 を追加します
# Load the library
library(qqman)
# Make the Manhattan plot on the gwasResults dataset
manhattan(gwasResults, chr="CHR", bp="BP", snp="SNP", p="P", col = c("#0057B7", "#FFDD00"), annotatePval = 0.01)
もう少し図にバリエーションを
3番染色体の集中していくつものSNPが高い-log10(p)値を示すのに頂上の変異しか名前が表示されないので annotateTop = FALSE というオプションをつけてみました。
# Load the library
library(qqman)
# Make the Manhattan plot on the gwasResults dataset
manhattan(subset(gwasResults, CHR == 3), chr="CHR", bp="BP", snp="SNP", p="P"
, col = c("#0057B7", "#FFDD00"), annotatePval = 0.001
, annotateTop = FALSE, main = "Chr 3")
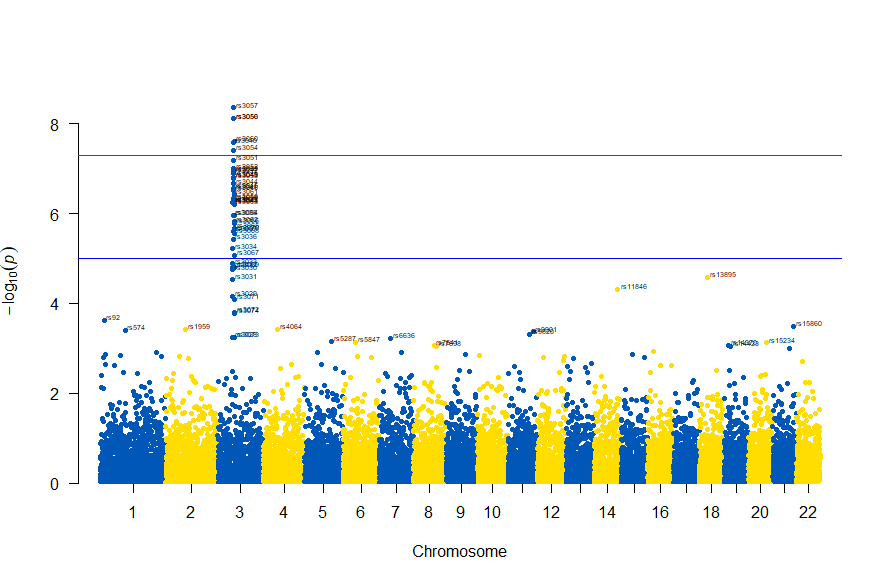
3番目の染色体に関連のありそうなSNPが集中しているようなので3番目のsubsetを表示してみます。肝は subset(gwasResults, CHR ==3) の個所です。
# Load the library
library(qqman)
# Make the Manhattan plot on the gwasResults dataset
manhattan(subset(gwasResults, CHR == 3), chr="CHR", bp="BP", snp="SNP", p="P"
, col = c("#0057B7", "#FFDD00"), annotatePval = 0.001
, annotateTop = FALSE, main = "Chr 3")
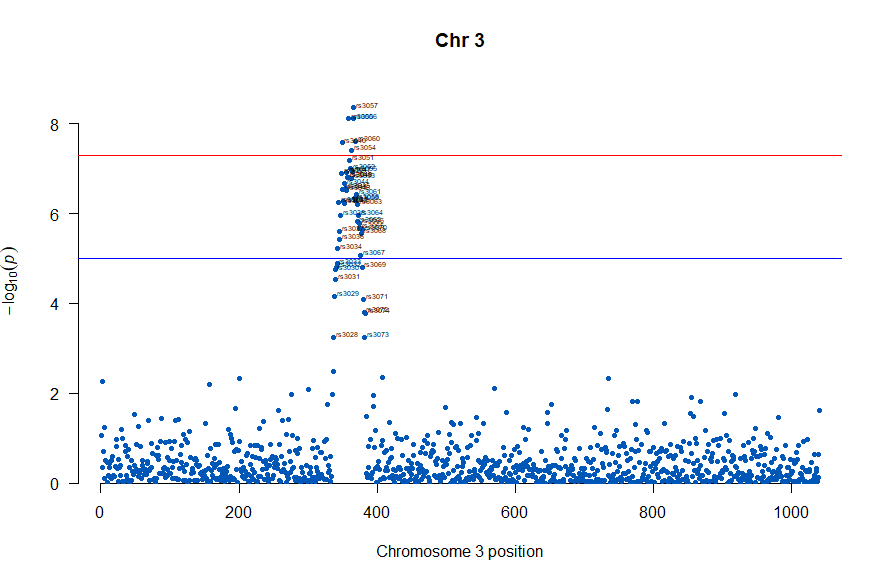
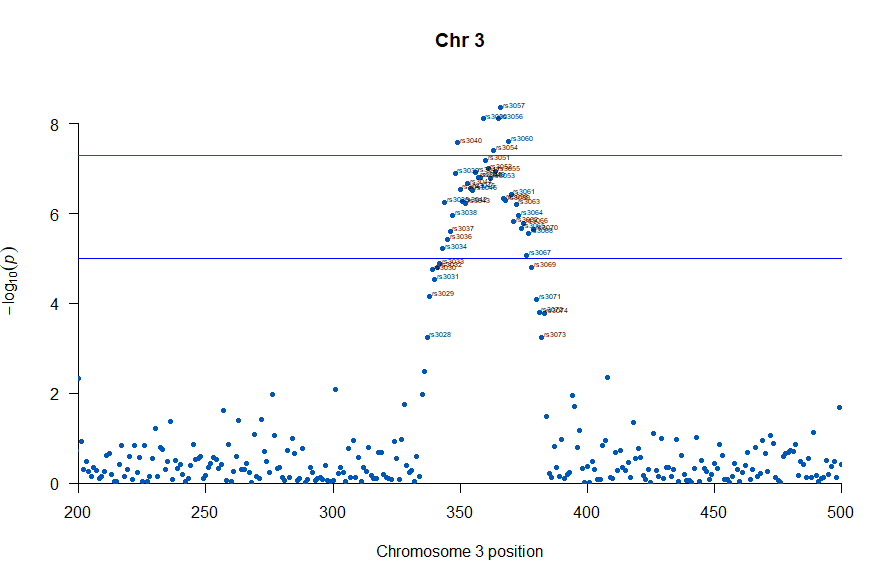
気になるあたりをもう少し拡大します
# Load the library
library(qqman)
# Make the Manhattan plot on the gwasResults dataset
manhattan(subset(gwasResults, CHR == 3), chr="CHR", bp="BP", snp="SNP", p="P"
, col = c("#0057B7", "#FFDD00"), annotatePval = 0.001
, annotateTop = FALSE, main = "Chr 3", xlim = c(200,500))
データの作成
上記の作図は単純で、各Chromosome上のそれぞれの位置でp値をプロットしただけのものなので、結局はp値を計算して大きな外れ値(-> association)を示す位置を目視で確認しやすくしたものだという事がわかりました。
肝となるのはp値の計算になります。それでは、p値をどうやって計算していたかというと、複数人のSNPのデータと、それぞれのサンプル(の人)の興味のある表現型のデータを用いて表現型と無関係なSNPか表現型とassociateしているSNPかを計算しています。
plink.exe (windows用)がインストールされていて、冒頭の書籍の付録のファイル 1KG_EUR_QC.bed, 1KG_EUR_QC.bim, 1KG_EUR_QC.fam とかのデータがあったとして、コマンドプロンプトで次を実施しました。
plink.exe --bfile 1KG_EUR_QC --out 1KG_QC_Pheno1 --pheno phenotype1.txt --logistic --ci 0.95
上記で作成されるファイルから、p値等のデータを抽出するのにawkを使いました。awkを使うのに、MacOSだとawkはデフォルトで入っているのですが、今回はwindowsを使いましたのでCYGWINで実行しました。
awk '{print $2"\t"($1*100000000000+$3)"\t"$12}' 1KG_QC_Pheno1.assoc.logistic > 1KG_QC_Pheno1.assoc.logistic.P.txt
この出力で生成される 1KG_QC_Pheno1.assoc.logistic.P.txt ファイルを上の方で記したRスクリプトのインプットとすることで、マンハッタンプロットを描くことができました。
公開データを使って練習
さらなる練習用という事で「心筋梗塞1666症例および対照健常者3198名のGWAS」データが公開されており、使用についての制限がないためこちらのデータで試してみました。
(制限がない=NBDC非制限公開データとは、アクセスに制限を設けることなく、誰でも利用することが可能な公開データです。)
次のサイトからダウンロードしました。
https://humandbs.biosciencedbc.jp/
「ゼロから実践する 遺伝統計学セミナー〜疾患とゲノムを結びつける」に従った例でawkで行っっていた、行の抽出の部分はをsqlで行いました。
好みの問題かもしれませんが、windowsでlinux emulatorみたいなのを稼働させてawk走らせるより、Microsoft AccessのSQLで抽出するのが素直に開発しやすいと感じました。
SELECT Release_L12vsUniv06_AllSample.[#SNPID] AS SNP, Release_L12vsUniv06_AllSample.chr AS chr, Release_L12vsUniv06_AllSample.chrloc AS location, Release_L12vsUniv06_AllSample.ptrend AS p INTO [0010 GWAS_results] FROM Release_L12vsUniv06_AllSample;
で作図は次のようにしました。
library(readxl)
library(qqman)
GWAS_results <- read_excel("0010 GWAS_results.xlsx")
manhattan(GWAS_results, chr="chr", bp="location", snp="SNP", p="p"
, col = c("#0057B7", "#FFDD00"), annotatePval = 0.00001
, annotateTop = FALSE)
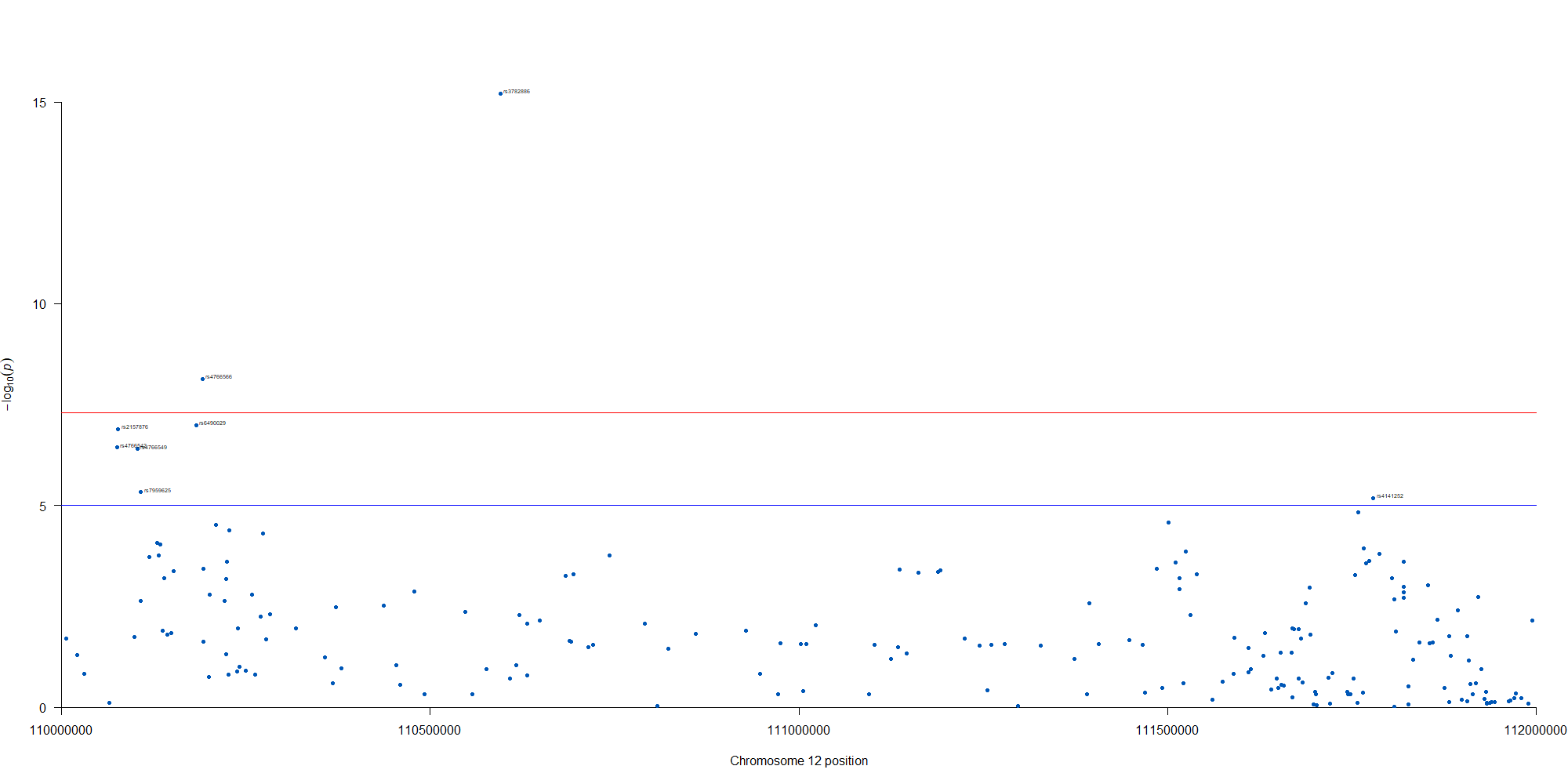
manhattan(subset(GWAS_results, chr == 12), chr="chr", bp="location", snp="SNP", p="p"
, col = c("#0057B7", "#FFDD00"), annotatePval = 0.00001
, annotateTop = FALSE, xlim = c(1.1e+8, 1.12e+08))
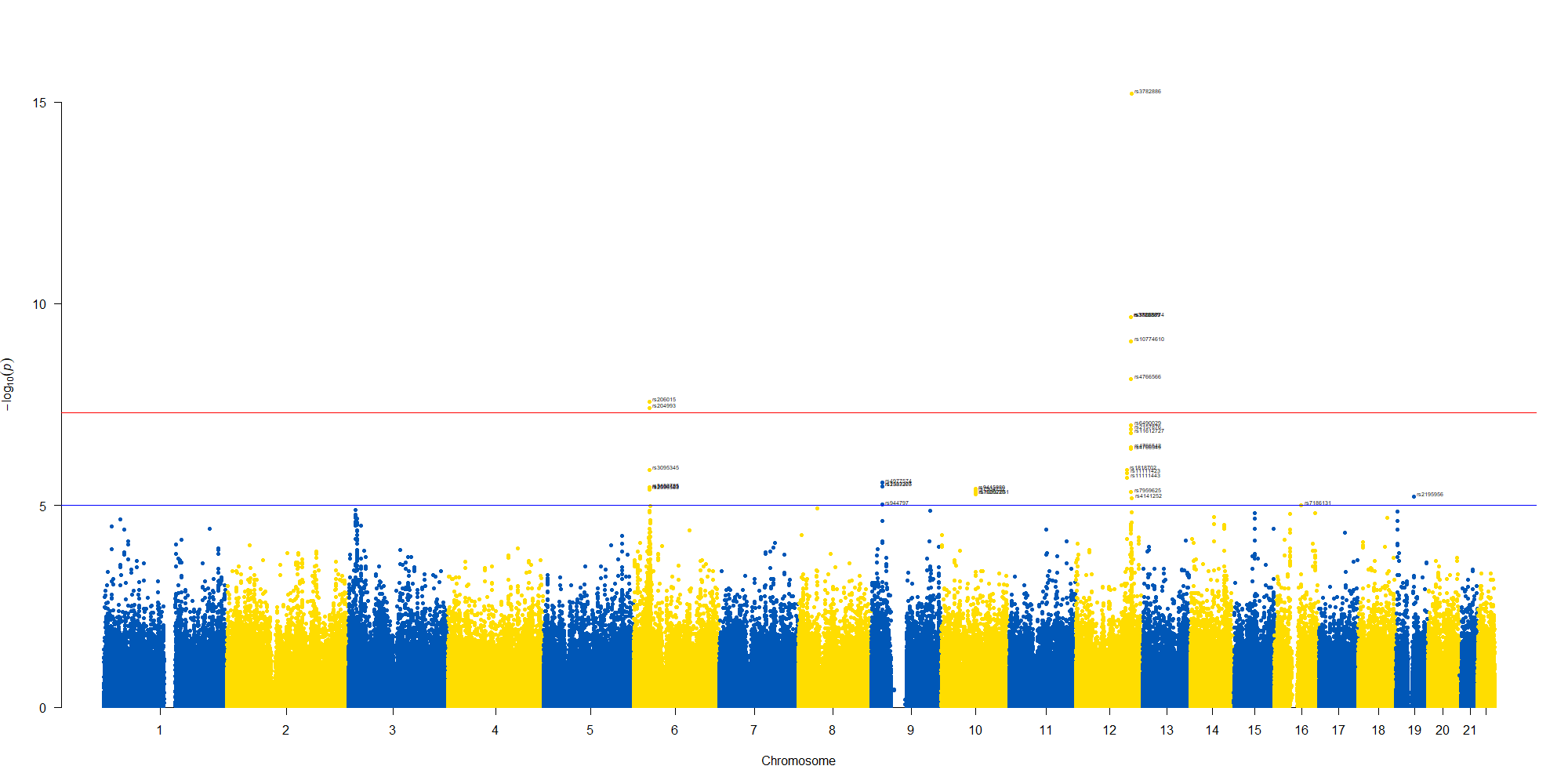
12番染色体の3’側に不均衡が集中している様なのでそのあたりを拡大したりしました。

公開データベースのアドレスが https://humandbs.dbcls.jp/ に変わっていますね。2024年4月から運用主体が変更になったとか。