機械学習的な何か
はじめに
その昔、2001年~2007年にかけて、遺伝子発現解析をやっていた頃、SVM (support vector machine)やNeural Networkを使用していました。肝臓由来の細胞に対し肝臓関連の副作用が知られている医薬品と、そうでない医薬品を曝露して、曝露後の遺伝子発現のパターンを見て、医薬品の肝毒性をin vitroで予測する、実験モデルを組み立てようとしていました。あまり、基礎的な経験のない中であれこれ考えても打開策が見つからず、また今思えば、根本的なデザインに無理があったようでもあり、結局なかなかよい出力が得られなかった苦い経験でした。
今回は基礎体力をつける意味で、インターネット上ですでにうまくいっているようなデータやスクリプトを基に自分なりに試してみます。正解があるものをトレースするのは、良い練習になります。
ナイーブベイズ分類器
今回試したのは、「ナイーブベイズ分類器」というものです。なぜこれを試すかと言うと、良い資料を見つけたからです。その資料を基に、試してみたという記事もあるようです。ざっと見たところ、数式でクラクラするのを我慢すると、何をやっているのかおぼろげながら見えてきます。あるカテゴリで相対的に高い頻度で出現する単語を指標にモデルを構築することになるようです。
スクリプト
機能する仕組みは使いながら考えるとして、スクリプトは次のようになります。全くリンク先の資料通りでは本当に芸がなさすぎるので、ちょっとだけ変えてみました。サンプルとしては、森鴎外と夏目漱石の作品を見分けるという課題で、MeCabのサイトで配布されているもの、を学習用の文章として、あとは青空文庫から「草枕」「こころ」(以上漱石)「花子」「あそび」(以上鴎外)をテスト用文章として、用いました。なお、青空文庫の文章はルビがうるさいので、delruby.exeを用いて処理したものでテストしました。
ルビの処理の様子(MS-DOSのコマンドプロンプトから)
delruby asobi.txt > ogai_asobi.txt
delruby hanako.txt > ogai_hanako.txt
delruby kokoro.txt > soseki_kokoro.txt
delruby kusamakura.txt > soseki_kusamakura.txt
これらの出力ファイルとMeCabのサイトから入手したデータをまとめて、setwdで指定したディレクトリの下の/data/writers/の下に置いて、次を実行しました。
library(RMeCab)
# data is downloaded from
# shift JIS
# http://web.ias.tokushima-u.ac.jp/linguistik/RMeCab/data.zip
# UTF8
# http://web.ias.tokushima-u.ac.jp/linguistik/RMeCab/data.tar.gz# indicate data folder
setwd(“C:/Users/Oshima/Documents/2018/R MeCab/”)# convert text files to vector
d <- t(docMatrix2(“data/writers”))myNaiveBayes <- function(x, y) {
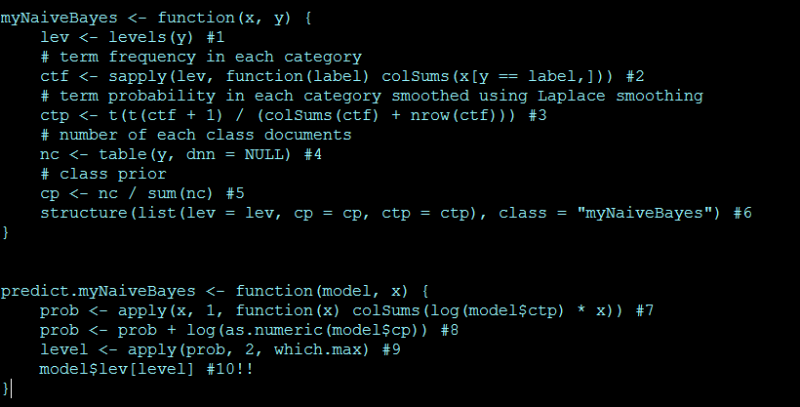
lev <- levels(y) #1
# term frequency in each category
ctf <- sapply(lev, function(label) colSums(x[y == label,])) #2
# term probability in each category smoothed using Laplace smoothing
ctp <- t(t(ctf + 1) / (colSums(ctf) + nrow(ctf))) #3
# number of each class documents
nc <- table(y, dnn = NULL) #4
# class prior
cp <- nc / sum(nc) #5
structure(list(lev = lev, cp = cp, ctp = ctp), class = “myNaiveBayes”) #6
}predict.myNaiveBayes <- function(model, x) {
prob <- apply(x, 1, function(x) colSums(log(model$ctp) * x)) #7
prob <- prob + log(as.numeric(model$cp)) #8
level <- apply(prob, 2, which.max) #9
model$lev[level] #10!!
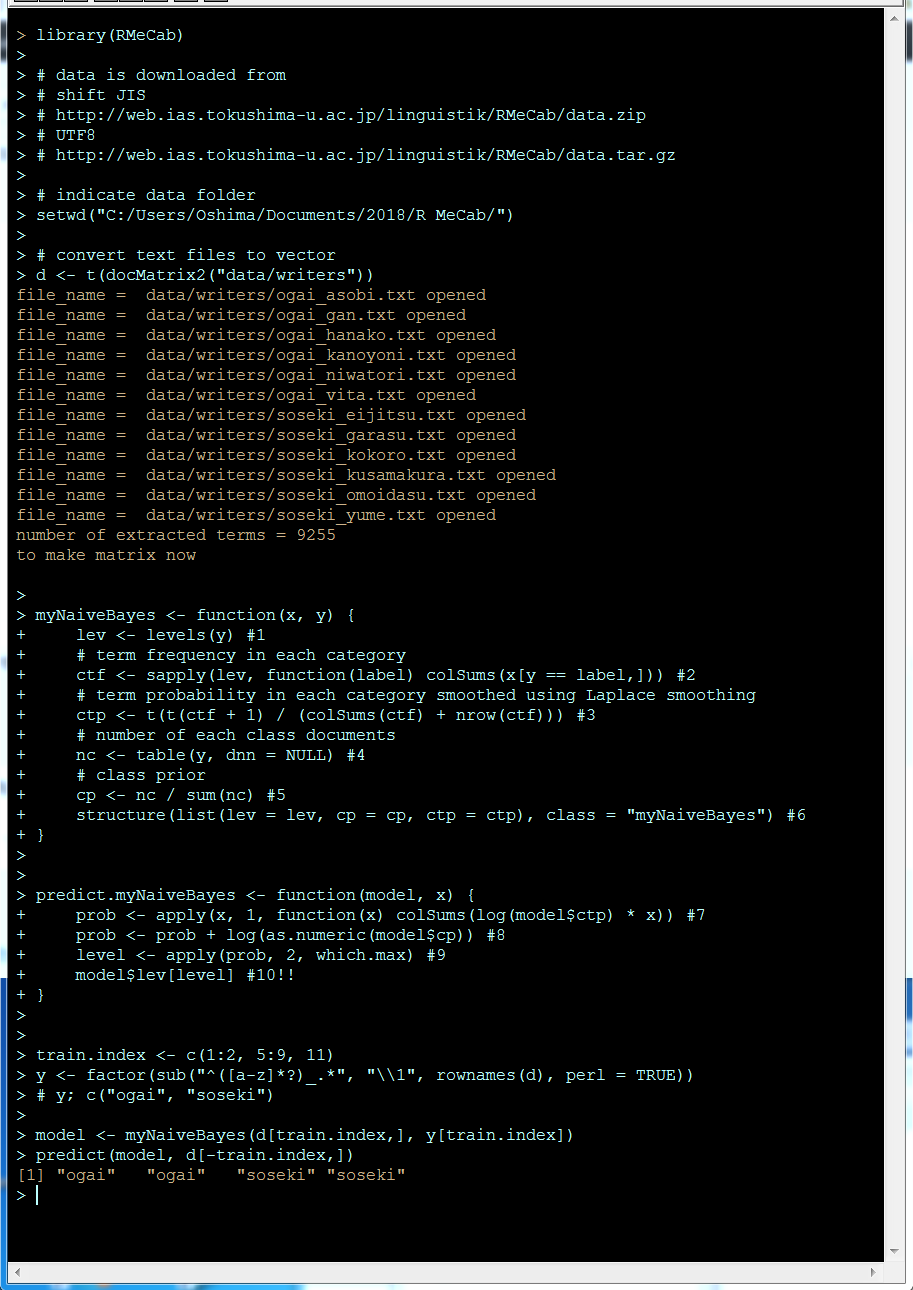
}train.index <- c(1:2, 5:9, 11)
y <- factor(sub(“^([a-z]*?)_.*”, “\\1”, rownames(d), perl = TRUE))
# y; c(“ogai”, “soseki”)model <- myNaiveBayes(d[train.index,], y[train.index])
predict(model, d[-train.index,])
ファイルは名前順にソートされますので、テストサンプルは、ogai, ogai, soseki, sosekiの順に出力されれば正解です。
おわりに
とりあえず、正解が得られていますが、これは元のリンク先の方の功績でしょう。正確な方法で他の手法と比較して実行時間を測定した訳ではないので印象なのすが、この手法は学習がかなり速いです。いずれにしても、とりあえずこのスクリプトの使い方は理解できたぞ。