1992年ころ、東京大学医科学研究所の白金通り[通称プラチナ通り]側の一角にピカッピカのプレハブの建物が建ちました。鳴り物入りで始まったヒトゲノム計画を担うヒトゲノム解析センターです。それまでは、医科学研究所では遺伝子解析にVMS社のVAXコンピュータで使用するGCGというソフトウェアが提供されていました。ヒトゲノム解析センターが稼働し始め、九州大学RIFISから鈴木さんという方がいらっしゃって、コンピュータの環境が変わってきました。ワークステーションがVMS VAXからSUN MicrosystemsのSun Sparc Stationに置き換わり、SUNの上でGCGが実行されるようになりました。そのプレハブの2階にはSUN以外にもいくつかのシステムが導入され、その中にSilicon Graphics社のIRISコンピュータというものがありました。そのIRIS上でQuantaというソフトウェアでは分子モデルを画面上に表示して、目で見ていろいろなことを考えることができました。それから二十数年コンピュータの進化は著しく、当時高額なワークステーションでしか実施できなかったような、分子モデルやドッキングが普通のパソコンでも実用的な時間内にできるようにっています。ということで、分子モデルの表示のプロセスの一部を忘備録として記載です。
- 使用するソフトウェアはSwiss-Pdb Viewerというフリーウェアで、開発にはGSK社の研究者らがかかわっていたと記憶しています。まずは、これを起動するとクレジットを表示する画面が現れます。この画面をクリックして先に進みます。
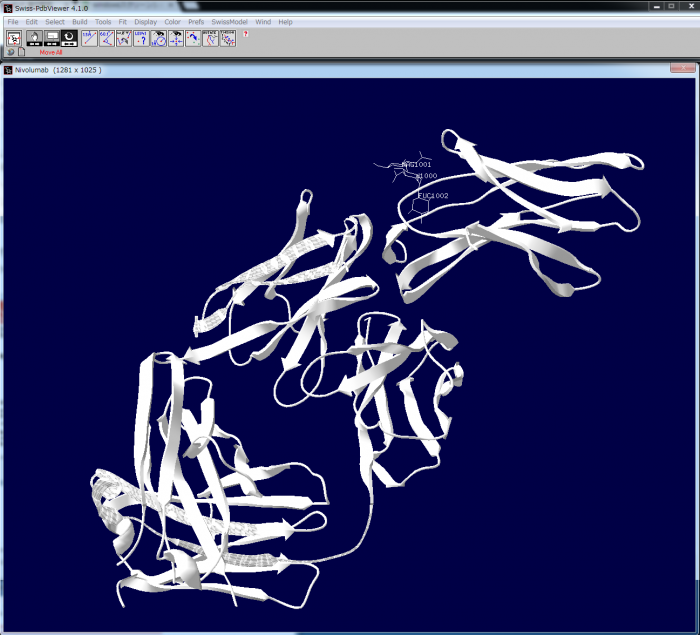
- メニューバーの <File> – <Open PDB File> から、目的のPDBファイルをロードします。データが取り込まれると、次のような画面になります。(設定によって、違う雰囲気の画面になることもあります。) 大画面のディスプレーにつないでいるときは1280×1024くらいのサイズで作業します。
- 分子サーフェスを見せたいので、<Tool> – <Surface> – <Compute> とします。計算されがサーフェスは設定していないと見えないので、計算が完了しても画面上は変化しないこともありますし、気持ちの悪い色のモコモコしたものが表示されることもあります。
- コントロールパネルの右上のcol BS (他のものになっていることもあります)の下向き三角形をクリックすると選択画面が現れますので、<molecular surface> を選びます。
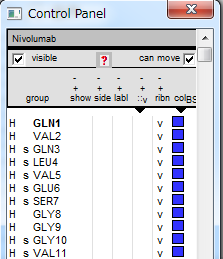
- 上のcontrol panelの中で青くなっている■をクリックして、好きな色に変更します。図では、灰色にしたところです。すると、分子表面の色も灰色になりました。
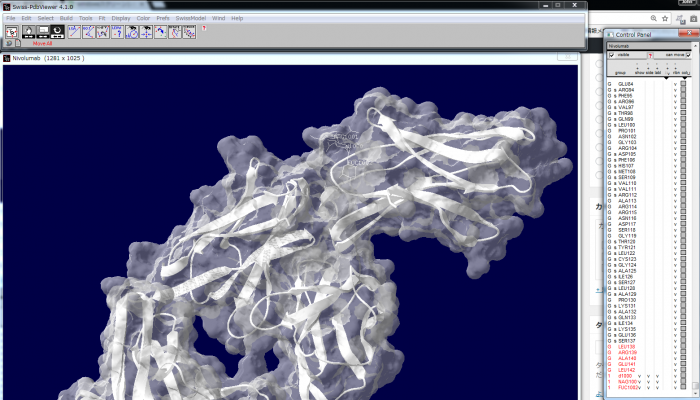
- アミノ酸残基ごとに好きな色に変更できます。私は、チェーンごとに色分けする事が多いです。荷電(陽性・陰性)を計算して色分けしてみたりもします(←コレで着想を得たこともあります)
- 出来上がったモデルをレンダリング用のファイルに書き出します。<File> – <Save> – <Povray scene>
- Povray で好きなバックグラウンドに浮かせて照明をあてて、好きな角度にカメラを置いてレイトレーシングすれば素敵な画になります。
