はじめに
こんな感じのデータがありました
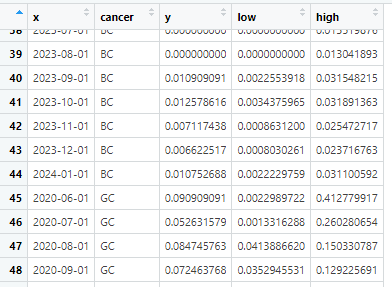
xは時期 as.Date(“2024-03-08”)とかの値が入っているベクター
cancer はc(“GC”, “BC”) がん種の値が入ったベクター
y は表示したい値(平均値とか頻度とか)
low/hight はエラーバーの上限・下限(95%信頼区間とか)
何がしたいのか
データフレームをがん種別に作成した後に、それらを一つのグラフで表示したくなった
library(ggplot2)
library(ggsci)
library(scales)
BC_data_frame <- data.frame(x, cancer, y, low, high) # BCとGCでそれぞれのベクターには
GC_data_frame <- data.frame(x, cancer, y, low, high) # 異なる値が入っている
combined_data_frame <- rbind(BC_data_frame, GC_data_frame)
g5 <- ggplot(combined_data_frame, aes(x, y, color = cancer)) +
geom_smooth(aes(ymin = low, ymax = high), alpha = 0.2) +
scale_color_nejm() +
geom_point() +
geom_errorbar(aes(ymin = low, ymax = high), width = 10) +
scale_x_date(limits = c(as.Date("2020-05-15"), as.Date("2024-01-31")), oob = oob_keep) +
scale_y_continuous(limits = c(0, 0.2), oob = oob_keep)
plot(g5)
ど
どうしたらどうなった
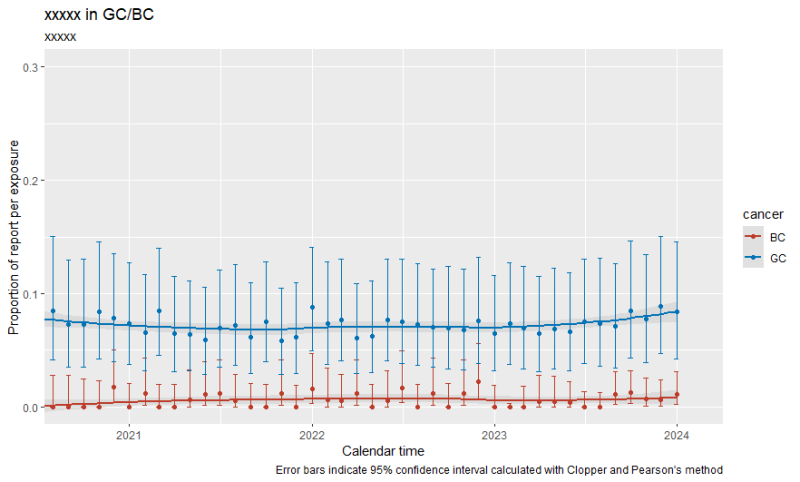
rbindで結合して、そのままプロットしたら2系列が分かれて表示された
全体はこんな感じ
setwd("C:/Users/****")
library(ggplot2)
library(ggsci)
library(scales)
x <- seq(as.Date("2020-06-01"), as.Date("2024-01-01"), by = "month")
k1 <- binom.test(1, 11); y <- c(k1$estimate); low <- c(k1$conf.int[1]); high <- c(k1$conf.int[2])
k2 <- binom.test(1, 19); y <- c(y, k2$estimate); low <- c(low, k2$conf.int[1]); high <- c(high, k2$conf.int[2])
# ... データ個所は中略
k43 <- binom.test(12, 135); y <- c(y, k43$estimate); low <- c(low, k43$conf.int[1]); high <- c(high, k43$conf.int[2])
k44 <- binom.test(11, 131); y <- c(y, k44$estimate); low <- c(low, k44$conf.int[1]); high <- c(high, k44$conf.int[2])
cancer <- rep("GC", 44)
GC_data_frame <- data.frame(x, cancer, y, low, high)
k1 <- binom.test(0, 12); y <- c(k1$estimate); low <- c(k1$conf.int[1]); high <- c(k1$conf.int[2])
k2 <- binom.test(0, 118); y <- c(y, k2$estimate); low <- c(low, k2$conf.int[1]); high <- c(high, k2$conf.int[2])
# ... データ個所は中略
k43 <- binom.test(2, 302); y <- c(y, k43$estimate); low <- c(low, k43$conf.int[1]); high <- c(high, k43$conf.int[2])
k44 <- binom.test(3, 279); y <- c(y, k44$estimate); low <- c(low, k44$conf.int[1]); high <- c(high, k44$conf.int[2])
cancer <- rep("BC", 44)
BC_data_frame <- data.frame(x, cancer, y, low, high)
combined_data_frame <- rbind(BC_data_frame, GC_data_frame)
g5 <- ggplot(combined_data_frame, aes(x, y, color = cancer)) +
geom_smooth(aes(ymin = low, ymax = high), alpha = 0.2) +
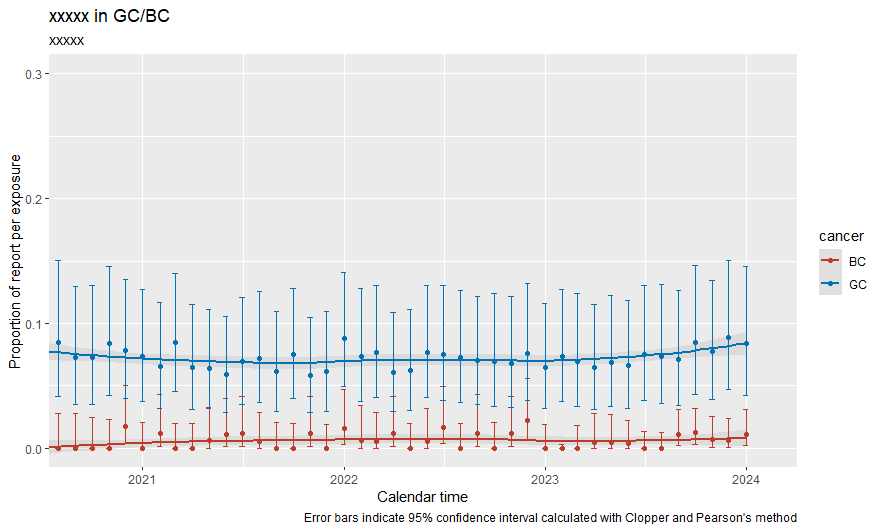
labs (title = "xxxxx in GC/BC") +
labs(subtitle = "xxxxx") +
labs( x = "Calendar time",
y = "Proportion of report per exposure",
caption = "Error bars indicate 95% confidence interval calculated with Clopper and Pearson's method" ) +
scale_color_nejm() +
geom_point() +
geom_errorbar(aes(ymin = low, ymax = high), width = 10) +
scale_x_date(limits = c(as.Date("2020-09-15"), as.Date("2024-02-01")), oob = oob_keep) +
scale_y_continuous(limits = c(0, 0.3), oob = oob_keep)
plot(g5)